拿下诺贝尔化学奖,类谷歌AlphaFold开源蛋白质大模型
添加书签
专注AIGC领域的专业社区,关注微软&OpenAI、百度文心一言、讯飞星火等大语言模型(LLM)的发展和应用落地,聚焦LLM的市场研究和AIGC开发者生态,欢迎关注!
本周谷歌DeepMind联合创始人兼首席执行官Demis Hassabis凭借AlphaFold系列模型拿下诺贝尔化学奖,创造了AI大模型首次拿下诺奖的历史。
尤其是最新发布的AlphaFold-3在生物分子结构、蛋白-配体结构、生物复合体等方面获得了很大提升,可显著加速人类对新药物的研发进程。
但AlphaFold-3模型是闭源的,并且在使用方面对于一些国家/地区也存在限制。所以,AI研究团队Chai开源了类似的多模态分子结构预测模型Chai-1。
开源地址:https://github.com/chaidiscovery/chai-lab/
在线地址:https://lab.chaidiscovery.com/auth/login?

虽然最近两年很多大模型也能预测蛋白质结构,但在多聚体预测或蛋白质 – 配体相互作用等方面的表现并不理想。
这是因为蛋白质多聚体的结构和相互作用非常复杂,涉及到多个蛋白质分子之间的精确排列和相互作用。使得模型在处理这种复杂性时可能存在困难,难以准确捕捉到多聚体中各个蛋白质分子之间的微妙相互作用和空间关系。
例如,多聚体中蛋白质分子的相对取向、结合位点的精确位置以及分子间的动态变化等因素对于其功能和活性至关重要,但现有的大模型可能无法充分考虑这些因素,导致预测结果不够准确。
而Chai-1的模型架构充分考虑了蛋白质多聚体的复杂性,使用了多种先进的技术来提高对多聚体结构和相互作用的预测能力。
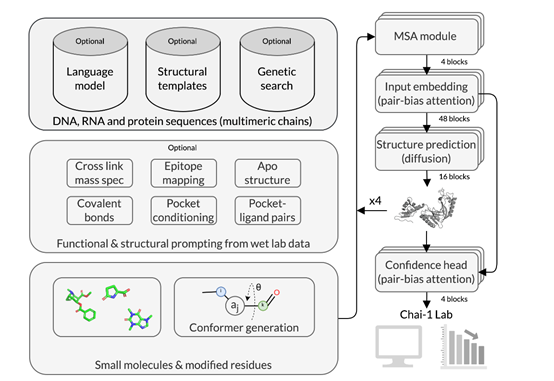
在模型架构方面,Chai – 1 通过引入来自大型蛋白质语言模型的残基级嵌入,能够更好地捕捉蛋白质序列中的信息,从而提高在单序列预测方面的能力。使得模型在处理缺乏多序列比对信息的情况时,仍然能够准确预测蛋白质的结构。
Chai – 1 还添加了约束特征,包括口袋约束、接触约束和对接约束等。这些约束特征能够模拟实验中观察到的蛋白质与其他分子之间的相互作用,帮助模型更准确地捕捉多聚体中各个蛋白质分子之间的微妙相互作用和空间关系。
例如,口袋约束可以指定蛋白质口袋与配体之间的距离限制,接触约束可以模拟蛋白质分子之间的接触情况,对接约束可以帮助模型确定蛋白质分子的相对取向。

此外,Chai – 1 在处理多聚体中蛋白质分子的相对取向时,能够利用其强大的计算能力和优化算法,对各种可能的取向进行全面的搜索和评估,从而找到最优的取向组合。
对于结合位点的精确位置,Chai – 1 通过对大量训练数据的学习,能够识别出微小的结构特征和化学信号,从而更准确地确定结合位点的位置。
同时,Chai – 1 还能够模拟分子间的动态变化,考虑了蛋白质分子的柔性和运动性,通过引入动态模型或采用机器学习算法来捕捉分子间的动态相互作用,从而更好地预测多聚体在不同条件下的结构和功能。
根据测试数据显示, Chai – 1 仅根据蛋白质序列和配体的化学组成,实现了 77%的配体 RMSD 成功率,与 AlphaFold3的76%相当;
在一个包含268个界面、跨越129个结构的评估集中,Chai-1无论是使用或不使用多重序列比对,在单序列模式下在抗体-蛋白的预测上显著优于AlphaFold 2.3。这表明Chai-1在处理抗体这类高度变异的免疫蛋白质序列时快速且准确率非常高。
本文素材来源Chai – 1 论文,如有侵权请联系删除
END


本篇文章来源于微信公众号: AIGC开放社区






