开源多结构蛋白质预测大模型——Genie 2
添加书签
专注AIGC领域的专业社区,关注微软&OpenAI、百度文心一言、讯飞星火等大语言模型(LLM)的发展和应用落地,聚焦LLM的市场研究和AIGC开发者生态,欢迎关注!
蛋白质结构主要由折叠成三维形状的氨基酸组成,想要设计具有精确结构和功能特性的蛋白质仍然有很大的技术难题,例如,酶催化或分子识别,这在各种生物和工业应用中至关重要。
为了帮助医疗、生物领域的研究人员提升对蛋白质的设计效率,哥伦比亚大学和罗格斯大学的研究人员联合开源了一款类似谷歌AlphaFold的蛋白质预测模型Genie 2。
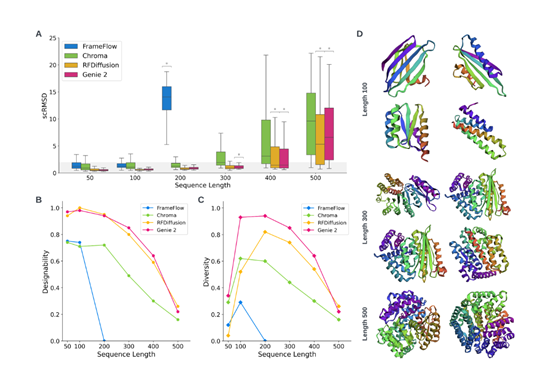
Genie 2是在Genie基础之上研发而成,不仅扩展了可设计蛋白质结构空间的广度和多样性,还引入了多模态能力,在蛋白质的预测准确率方面比现有同类模型Chroma、FrameFlow和RFDiffusion等更好。
开源地址:https://github.com/aqlaboratory/genie2
论文地址:https://arxiv.org/abs/2405.15489

技术创新方面,Genie 2采用了一种全新的条件生成方法,这种方法允许模型在生成过程中考虑特定的序列和结构信息。
Genie 2将每个motif的残基编码为一位有效向量,并将这些编码与单个残基特征结合起来,从而将条件信息整合到扩散过程中。
这种巧妙的编码方法,能够以SE(3)不变的方式来编码主题结构。也就是说,模型不会对主题的绝对位置和方向敏感,这大大提高了设计的灵活性和鲁棒性。
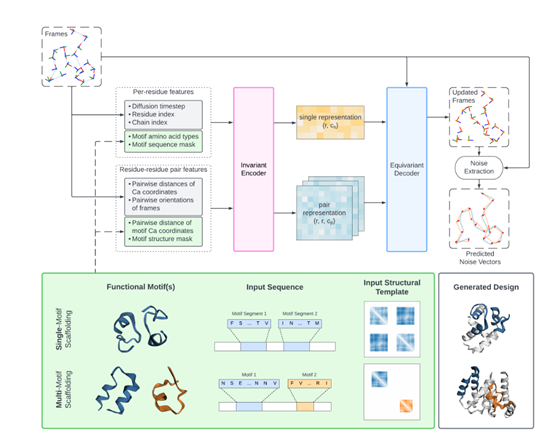
为了使Genie 2能够捕捉到复杂的蛋白质结构分布,研究人员使用了大规模数据增强方法,就是使用目前全球最大的蛋白质数据库谷歌的AlphaFold数据库。
这是一个包含约2.14亿个AlphaFold系列模型预测的大型数据库,几乎覆盖了UniProt数据库中的所有蛋白质。这种大规模的数据增强,使得Genie 2能够学习到比以往任何模型都要丰富的蛋白质结构信息,在预测和解构方面也更加精准。
但并非所有AlphaFold中的数据都适用于训练。研究人员为了提炼高质量数据,使用了FoldSeek对数据库中的条目进行了结构相似性聚类,并设置了pLDDT阈值大于80和最大序列长度为256,以筛选出高质量的蛋白质预测结构。
在训练Genie 2的过程中,研究人员使用了一种特定的损失函数,该函数计算预测噪声和真实噪声之间的均方误差。这种损失函数的设计使得模型在生成蛋白质结构时,能够更加关注于满足主题的约束条件,同时保持对整体蛋白质设计的响应性。
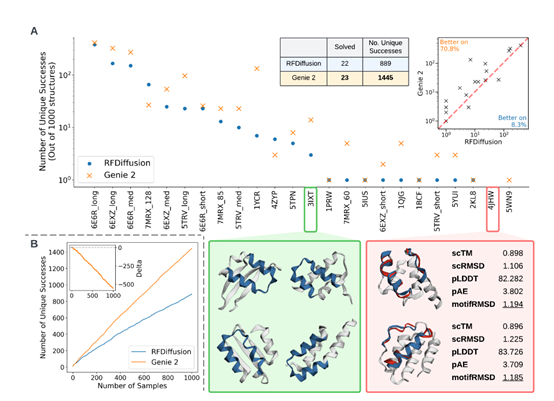
Genie 2的性能在一系列无条件和条件生成任务中接受了严格测试和评估,结果显示,其在设计能力、多样性及创新等关键指标上均超越Chroma、FrameFlow和RFDiffusion知名模型。
尤其是在多模体支架构建任务上,Genie 2不仅解决了更多问题,而且提出了更多样化且独特的解决方案。
研究团队精心设计了一个包含6个多模体支架构建问题的基准集,涵盖了从免疫原到结合剂再到酶设计的各种潜在蛋白质设计任务,Genie 2成功解决了其中的4项任务。例如,成功设计了包含四个钙离子结合位点的支架以及整合了RSV-F site II和RSV-G 2D10表位的复杂结构。
本文素材来源Genie 2论文,如有侵权请联系删除
END

本篇文章来源于微信公众号: AIGC开放社区






